Piattaforma di Informatica Molecolare
Descrizione
Competenze
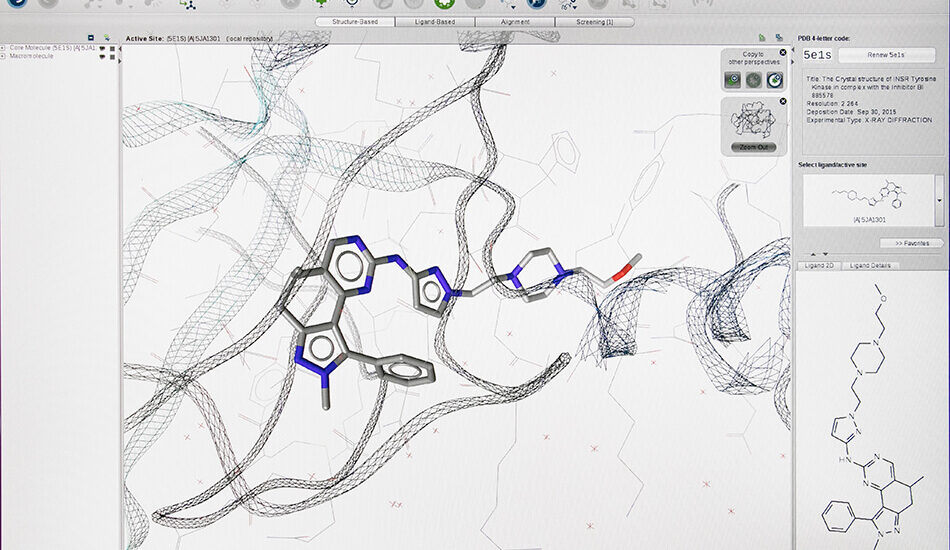
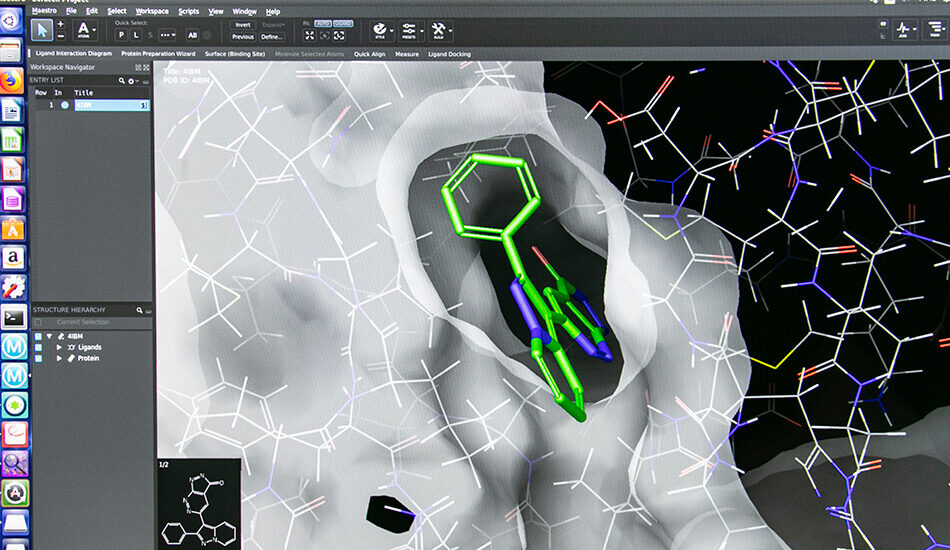
- Virtual screening structure-based (Docking e Approccio farmacoforico);
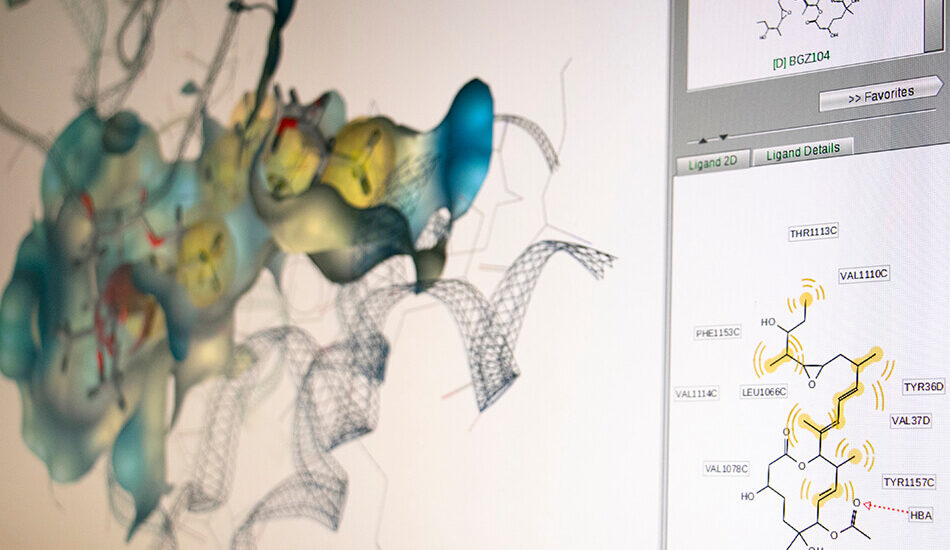
- Virtual screening ligand-based (farmacoforo, modelli basati su descrittori molecolari, QSAR e 3D QSAR);
- Dinamica molecolare;
- Farmacoforo dinamico (tecnica ibrida basata sull’utilizzo di farmacofori generati sulle traiettorie di dinamica molecolare);
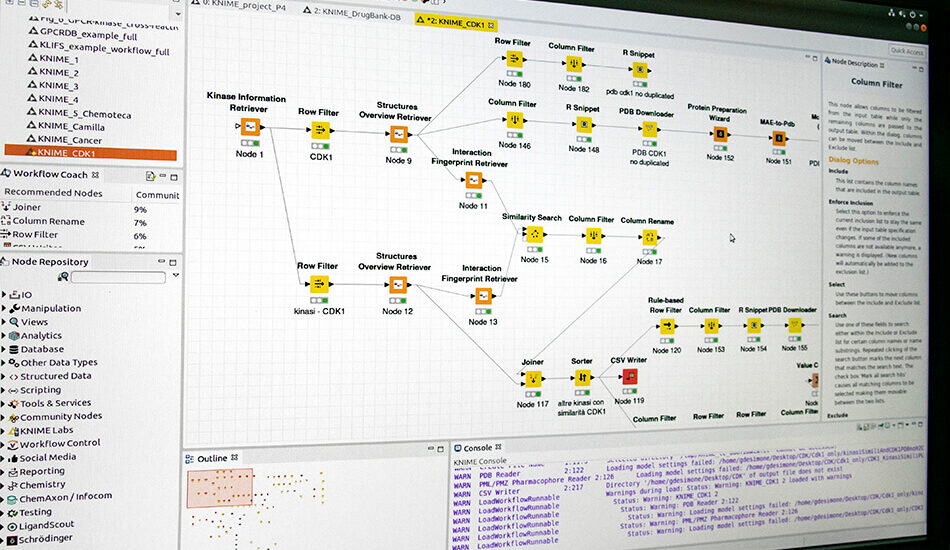
- Creazione e gestione di Database molecolari;
- Data mining applicato a problematiche legate alla chimica medicinale;
- Neural Network in Drug Design.
Dotazione tecnologica
Software
-
- Schrödinger suite for small molecule drug discovery
- Schrödinger suite for biologics drug discovery
- LigandScout expert suite
- Autodock and Autodock Vina
- AlvaDesc/AlvaModel
- DESMOND (OPLS2005 and OPLS3e, OPLS4)
- AMBER
- NAMD
- VMD
- GROMACS
- KNIME
Piattaforma integrata in silico
Il gruppo ha realizzato la piattaforma integrata per lo studio dei network molecolari (OBIND) in collaborazione con il gruppo di bioinformatica.
Contatti:
Collaborazioni:
- University of Vienna (Pharmaceutical Chemistry Dep.), Austria
- Universitè de Paris Cité, France
- Università di Napoli Federico II, Italy
- Univeristà degli Studi di Verona, Italy
- Consiglio Nazionale delle Ricerche IFT-CNR, Palermo, Italy
- Univeristà degli Studi di Palermo, Italy
Hardware
Hardware
- 6 Workstations
- Server: 80 cores e 2 x NVIDIA Tesla K80
- Server: 96 cores e 2 x NVIDIA A100
Capacità di calcolo:
- Library optimisation → ~ 6,000 molecole/min
- Virtual Screening HTVS → ~ 5,000 molecole /min
- Virtual Screening SP → ~ 1,500 molecole /min
- Molecular Dynamics → ~ 200 ns/giorno/scheda (su un sistema medio di 40,000 atomi)