Sviluppo di nuovi strumenti per la lotta al SARS-CoV-2
Abstract
La pandemia di COVID-19 ha portato allo sviluppo di diverse strategie per prevenire l’infezione o alleviarne le manifestazioni cliniche. Finora, la maggior parte degli sforzi è stata investita nello sviluppo di vaccini, in grado di indurre una risposta immunitaria contro la proteina virale Spike, e di anticorpi neutralizzanti la Spike. Tuttavia, considerando la natura mutante del SARS-CoV-2 che interessa principalmente la regione Spike, l’efficacia degli strumenti finora disponibili non è stata dimostrata per le varianti attuali e future del virus.
Inoltre, l’infezione da SARS-CoV-2 altera l’omeostasi delle cellule infette, utilizzando il macchinario replicativo della cellula per completare il ciclo di vita del virus. È stato scoperto che diverse proteine ospiti sono coinvolte nelle interazioni proteina-proteina con il complesso di trascrizione e replicazione virale (RTC), composto da tre proteine virali non strutturali (nsps): nsp12 (RNA polimerasi RNA-dipendente), che media la polimerizzazione nucleotidica, e nsp8 e nsp7, che agiscono come primasi. In particolare, precedenti risultati basati su studi di proteomica e modelli computazionali hanno suggerito che le proteine che legano l’RNA, come le La-Related Proteins (LaRPs), una superfamiglia di proteine caratterizzate da un motivo N-terminale estremamente conservato (modulo La), sono legate alla regolazione degli RNA cellulari e potrebbero essere coinvolte nella replicazione del genoma virale.
I nostri studi sono focalizzati da un lato sulla caratterizzazione biofisica e strutturale delle possibili interazioni che coinvolgono la triade nsp12-8-7 dell’RTC con i fattori della cellula ospite LaRPs, per determinare se uno o più membri di questa peculiare superfamiglia sia cruciale per la replicazione virale. Fornendo informazioni cinetiche e strutturali, utilizzando tecniche strutturali e biofisiche, come Bio-Layer Interferometry (BLI), Isothermal Titration Calorimetry (ITC), la Risonanza magnetica nucleare (NMR) e la crio-microscopia elettronica (cryo-EM), la nostra ricerca intende contribuire allo sviluppo di nuovi approcci terapeutici.
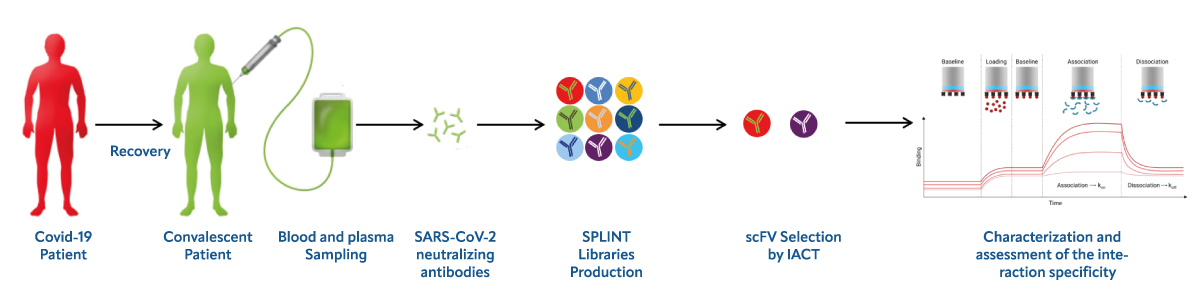
Dall’altro lato, l’obiettivo del nostro progetto di ricerca è quello di trovare trattamenti più duraturi e ad ampio spettro che siano efficaci contro le numerose varianti del virus, e che possano anche contrastare future pandemie da coronavirus. In particolare, siamo impegnati nello sviluppo di anticorpi, sotto forma di single chain fragment variable (scFv), contro la proteina del Nucleocapside (N) e la proteina Nsp9 del SARS-CoV-2, piuttosto che sulla sola proteina Spike, la cui rapida ed elevata mutabilità comporta una riduzione dell’efficacia dei trattamenti disponibili. Entrambe le proteine scelte sono di estrema rilevanza biologica e terapeutica: Nsp9 è una proteina dimerica legante ssRNA altamente conservata tra i Betacoronavirus ed è coinvolta nella replicazione e nella trascrizione del virus; la proteina N, anch’essa dimerica, impacchetta il filamento positivo dell’RNA virale e svolge un ruolo importante nel migliorare l’efficienza di trascrizione dell’RNA virale subgenomico.
Impatto
Dall’inizio della pandemia di COVID-19, la maggior parte degli sforzi è stata investita nello sviluppo di vaccini in grado di indurre una risposta immunitaria contro la proteina virale Spike, e di anticorpi neutralizzanti la Spike. Tuttavia, una delle domande a cui i ricercatori hanno subito cercato di rispondere riguarda l’estrema variabilità del virus SARS-CoV-2, dovuta principalmente alla natura altamente mutabile della proteina Spike, che rende gli strumenti terapeutici sviluppati finora sempre meno efficaci. Pertanto, rimane essenziale indagare su altre fasi dell’infezione e della replicazione virale, nonché sul meccanismo molecolare con cui i virus rapiscono e sfruttano i fattori dell’ospite. In questo contesto, una conoscenza più approfondita del ruolo che le proteine dell’ospite svolgono durante la replicazione virale contribuirà a capire le basi molecolari dell’infezione dei coronavirus.
Il nostro obiettivo è anche quello di sviluppare nuove molecole contro proteine diverse da Spike, implementando al contempo una piattaforma e un flusso di lavoro per un intervento rapido e decisivo in caso di future pandemie di coronavirus. I dati prodotti finora su Nsp9 e sulla proteina del Nucleocapside forniscono il prerequisito per selezionare e sviluppare nuovi anticorpi neutralizzanti contro queste due proteine, impedendo così la replicazione virale e/o attenuando l’aggressività del virus.
Pipeline
-
CLINICAL
NEED -
DISEASES
ANALYSIS - DISCOVERY
-
PRECLINICAL
VALIDATION -
PRECLINICAL
DEVELOPMENT -
CLINICAL
STUDIES
Principal Investigator
Contact
Therapeutic Areas:
Product:
Drugs – Biologics
Collaborations:
- Randall Division of Cell and Molecular Biophysics, King’s College London (KCL), Regno Unito
- European Brain Research Institute Rita Levi Montalcini (EBRI) – Roma, Italia
- International Covid-19 NMR Consortium
Scarica il pdf del progetto