Sviluppo di strumenti per l’analisi di dati ottenuti da sequenziamento dell'RNA a cellula singola
Abstract
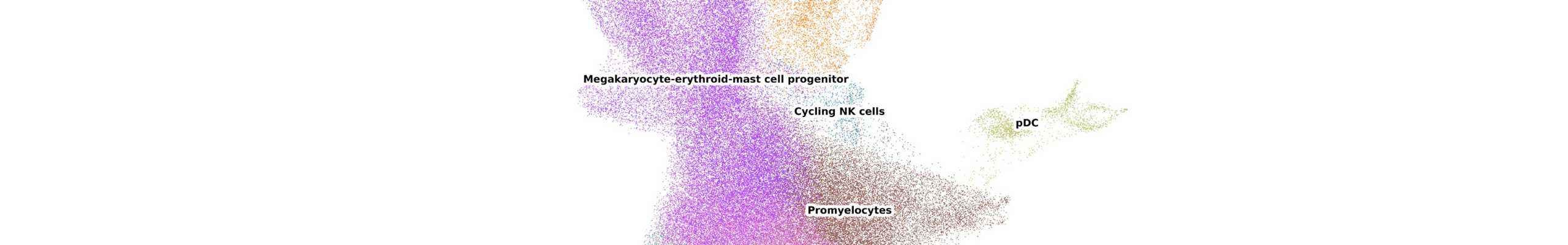
Il sequenziamento dell’RNA a singola cellula (scRNA-seq) consente di analizzare l’espressione genica a livello di una singola cellula. Rispetto all’approccio standard del sequenziamento bulk (RNA-seq), il scRNA-seq consente di caratterizzare ciascuna cellula a livello del trascrittoma, aprendo alla possibilità di identificare popolazioni cellulari rare ma funzionalmente importanti. Inoltre, il scRNA-seq è uno strumento potente che permette di scoprire nuove tipologie di cellule nei vari tessuti umani, fornendo informazioni più approfondite su difetti dello sviluppo, immunologia, Covid-19, cancro, diagnosi clinica e molti altri campi di studio.
I dati derivanti da scRNA-seq sono ad altà dimensionalità: per ogni campione, migliaia di cellule esprimono migliaia di geni. Tuttavia, un gene potrebbe essere espresso in una cellula, ma non essere espresso in un’altra cellula. A causa di questo effetto denominato “dropout”, i dati scRNA-seq sono spesso molto sparsi. Pertanto, il tradizionale approccio bioinformatico deve essere adattato e nuovi tool devono essere implementati per analizzare questo particolare tipo di dati. In questo contesto stiamo lavorando alla definizione e validazione di una pipeline di elaborazione che funzioni sia in ambiente Python che in ambiente R.
Se il campione sperimentale cela un processo biologico, come la differenziazione di cellule staminali, il scRNA-seq consente di ricostruire la dinamica temporale dell’espressione genica che caratterizza il lignaggio. Questo approccio prende il nome di analisi dello pseudotempo, ed è uno dei temi su cui stiamo lavorando, testando gli strumenti esistenti e sviluppando nuovi metodi statistici.
Questo progetto è supervisionato dalla Dott.ssa Nicolina Sciaraffa, Scientist in Biostatistica.
Pipeline
-
CLINICAL
NEED -
DISEASES
ANALYSIS - DISCOVERY
-
PRECLINICAL
VALIDATION -
PRECLINICAL
DEVELOPMENT -
CLINICAL
STUDIES
Principal Investigator
Contatto
Aree terapeutiche:
Prodotto:
Tool Bioinformatici
Collaborazioni:
Consiglio Nazionale delle Ricerche, Palermo, Italia
Università degli Studi di Palermo, Palermo, Italia
Scarica il pdf del progetto
