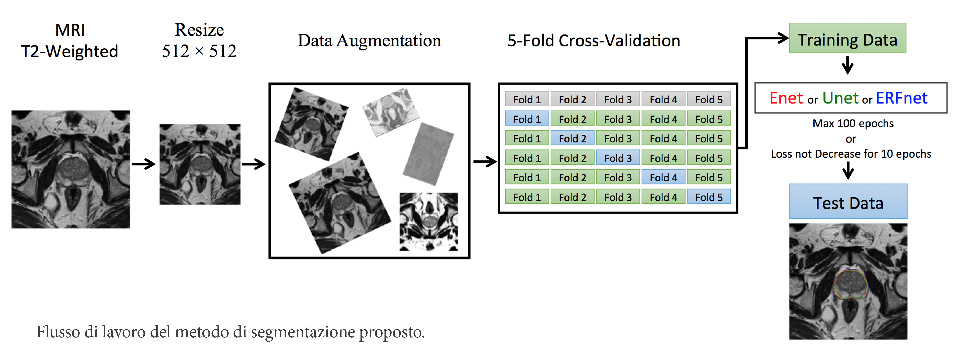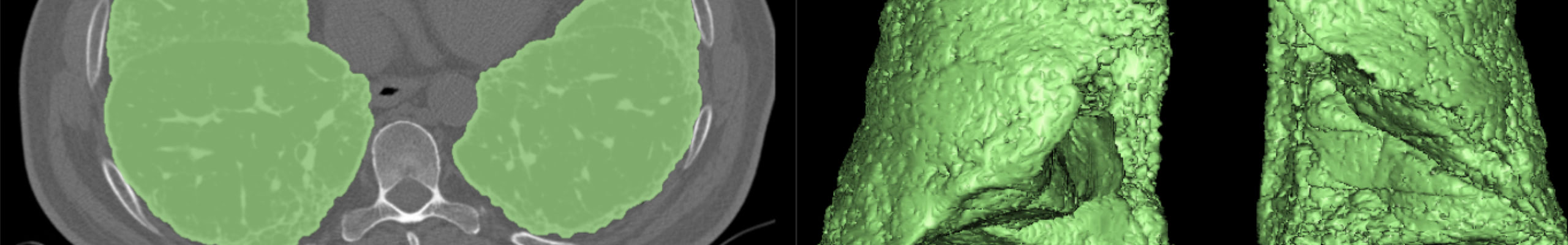
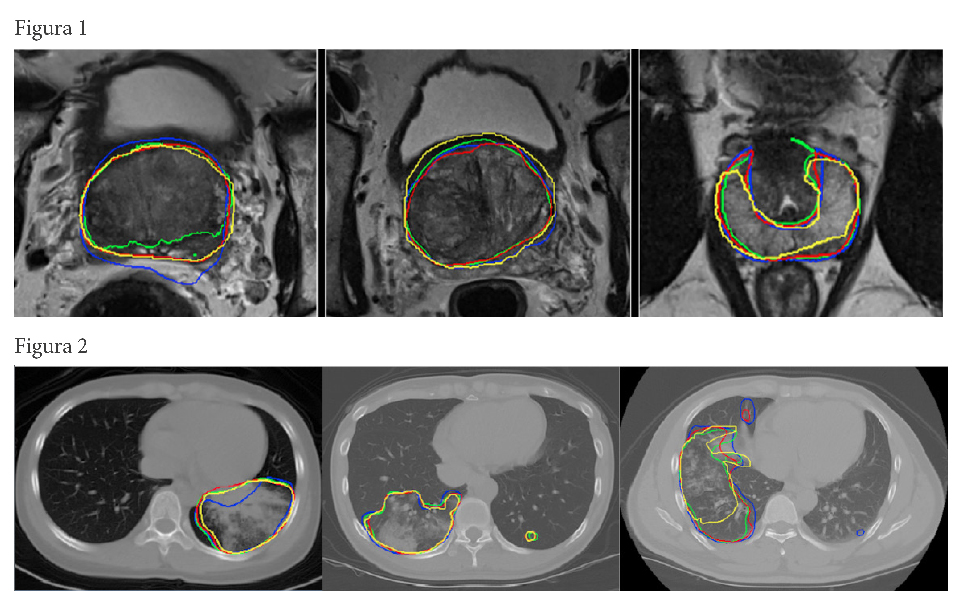
Sviluppo di algoritmi automatici 3D per la segmentazione e la classificazione di immagini biomediche
Abstract
Nel campo dell’imaging biomedico, la segmentazione del target viene abitualmente utilizzata come primo passo in qualsiasi sistema automatizzato di diagnosi delle malattie (o sistema di radioterapia) e, negli ultimi anni, negli studi di radiomica per ottenere grandi volumi di dati quantitativi da immagini mediche.
Questi dati vengono quindi utilizzati come biomarcatori di imaging per identificare qualsiasi possibile associazione con l’esito del paziente. Il primo passo di un flusso di lavoro radiomico è la delineazione del target (es. Tumore o organo) in modo tale da evitare distorsioni nell’estrazione dei parametri. Sebbene la delineazione manuale sembri il modo più intuitivo e facilmente implementabile per ottenere il volume target, è un processo che richiede tempo ed è soggetto alla massima variabilità inter e intra-osservatore.
Questa variabilità causa risultati irriproducibili nella firma radiomica che è fortemente influenzata dalla regione di interesse disegnata per identificare il tumore. Per questo motivo, è obbligatorio un metodo di delineazione degli obiettivi automatico e indipendente dall’operatore.
Impatto:
Il processo di segmentazione rimane un’area di ricerca popolare e stimolante. Oggigiorno l’attività clinica pone un elevato livello di domanda su algoritmi di segmentazione, necessari per produrre risultati ripetibili, indipendenti dalle scelte compiute dall’utente e capaci di elaborare in tempo reale. Il progetto propone un sistema di segmentazione appositamente ingegnerizzato per raggiungere il massimo livello di automazione e in grado di ottenere una segmentazione indipendente dall’operatore. Nello specifico, proponiamo due metodologie:
– Una superficie attiva (AS) completamente 3D guidata da un componente di apprendimento automatico 3D (ovvero classificazione dei tessuti 3D) per la segmentazione dei tumori nei distretti polmonari, della testa e del collo e del cervello in PET.
– Un framework di apprendimento profondo per la segmentazione dell’aorta aneurismatica e della sua valvola ma anche del parenchima con fibrosi polmonare idiopatica che fornisce risultati di segmentazione rapidi e precisi dopo essere stati addestrati con un set piccolo di immagini di CT ad alta risoluzione.
Pipeline
-
CLINICAL
NEED -
DISEASES
ANALYSIS - DISCOVERY
-
PRECLINICAL
VALIDATION -
PRECLINICAL
DEVELOPMENT -
CLINICAL
STUDIES
Principal Investigator
Contatto
Aree terapeutiche:
Prodotto:
Biomarcatori, Dispositivi biomedicali e Organi artificiali
Collaborazioni:
- Istituto Mediterraneo per i Trapianti (ISMETT) IRCCS, Palermo, Italia
- Istituto di Bioimmagini e Fisiologia Molecolare (IBFM-CNR), Cefalù, Italia
- Georgia Institute of Technology (GIT), Atlanta, USA
- Unità di Medicina Nucleare, Università di Messina, Messina, Italia
- Unità di Medicina Nucleare, Fondazione Istituto G.Giglio, Cefalù, Italia
- Dipartimento di Ingegneria, Università di Palermo, Palermo, Italia
- Unità di Fisica Medica, Ospedale Cannizzaro, Catania, Italia
- Dipartimento di Medicina Nucleare, Ospedale Cannizzaro, Catania, Italia
- Dipartimento di Medicina e Chirurgia Clinica, Università degli Studi di Napoli “Federico II”, Napoli, Italia
Scarica il pdf del progetto